來源:資料STUDIO作者:Jim大量資料中具有"相似"特徵的資料點或樣本劃分為一個類別。聚類分析提供了樣本集在非監督模式下的類別劃分。聚類的基本思想是"物以類聚、人以群分",將
2021-07-22 03:03:12
來源:資料STUDIO
作者:Jim
大量資料中具有"相似"特徵的資料點或樣本劃分為一個類別。聚類分析提供了樣本集在非監督模式下的類別劃分。聚類的基本思想是"物以類聚、人以群分",將大量資料集中相似的資料樣本區分出來,並發現不同類的特徵。
聚類模型可以建立在無類標記的資料上,是一種非監督的學習演算法。儘管全球每日新增資料量以PB或EB級別增長,但是大部分資料屬於無標註甚至非結構化。所以相對於監督學習,不需要標註的無監督學習蘊含了巨大的潛力與價值。聚類根據資料自身的距離或相似度將他們劃分為若干組,劃分原則是組內樣本最小化而組間距離最大化。

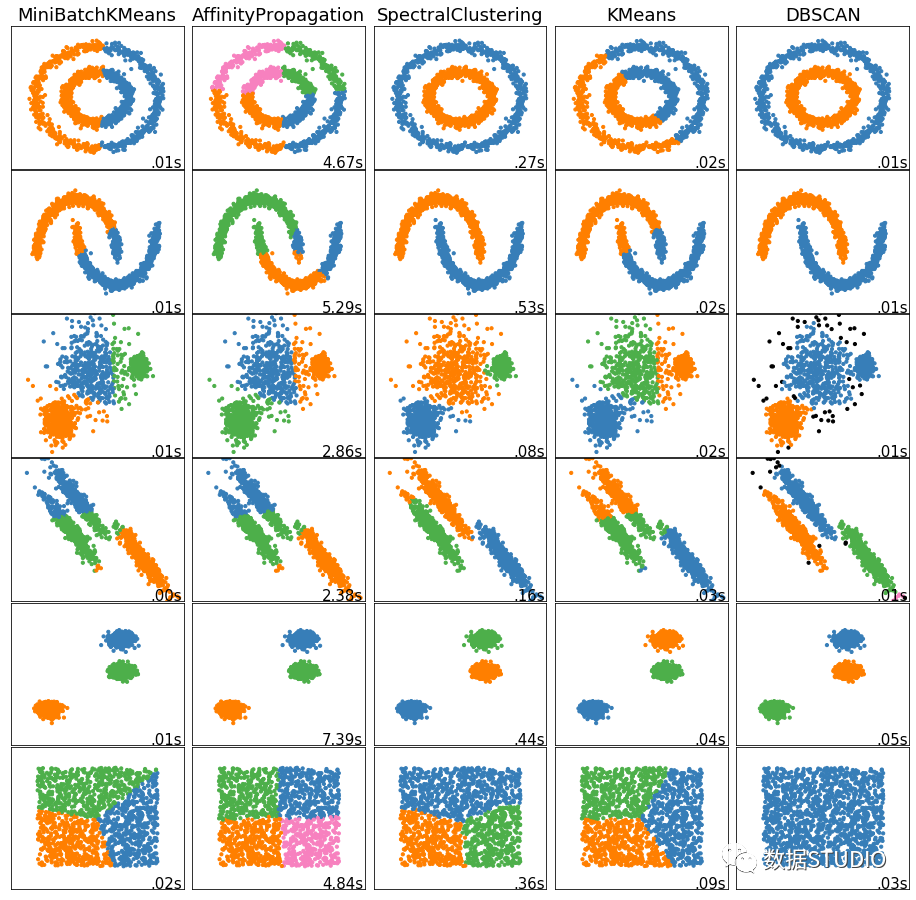
常見聚類演算法聚類效果對比圖
聚類分析常用於資料探索或挖掘前期
沒有先驗經驗做探索性分析
樣本量較大時做預處理
常用於解決
資料集可以分幾類;每個類別有多少樣本量
不同類別中各個變數的強弱關係如何
不同類型的典型特徵是什麼
一般應用場景
群類別間的差異性特徵分析
群類別內的關鍵特徵提取
影象壓縮、分割、影象理解
異常檢測
資料離散化
當然聚類分析也有其缺點
無法提供明確的行動指向
資料異常對結果有影響
本文將從演算法原理、優化目標、sklearn聚類演算法、演算法優缺點、演算法優化、演算法重要參數、衡量指標以及案例等方面詳細介紹KMeans演算法。
K均值(KMeans)是聚類中最常用的方法之一,基於點與點之間的距離的相似度來計算最佳類別歸屬。
KMeans演算法通過試著將樣本分離到 個方差相等的組中來對資料進行聚類,從而最小化目標函數 (見下文)。該演算法要求指定叢集的數量。它可以很好地擴展到大量的樣本,並且已經在許多不同領域的廣泛應用領域中使用。
被分在同一個簇中的資料是有相似性的,而不同簇中的資料是不同的,當聚類完畢之後,我們就要分別去研究每個簇中的樣本都有什麼樣的性質,從而根據業務需求制定不同的商業或者科技策略。常用於客戶分群、使用者畫像、精確營銷、基於聚類的推薦系統。
從 個樣本資料中隨機選取 個質心作為初始的聚類中心。質心記為
定義優化目標
開始迴圈,計算每個樣本點到那個質心到距離,樣本離哪個近就將該樣本分配到哪個質心,得到K個簇
對於每個簇,計算所有被分到該簇的樣本點的平均距離作為新的質心
直到 收斂,即所有簇不再發生變化。

KMeans迭代示意圖
KMeans 在進行類別劃分過程及最終結果,始終追求"簇內差異小,簇間差異大",其中差異由樣本點到其所在簇的質心的距離衡量。在KNN演算法學習中,我們學習到多種常見的距離 ---- 歐幾里得距離、曼哈頓距離、餘弦距離。
在sklearn中的KMeans使用歐幾里得距離:
則一個簇中所有樣本點到質心的距離的平方和為:
其中, 為一個簇中樣本的個數, 是每個樣本的編號。這個公式被稱為簇內平方和(cluster Sum of Square), 又叫做Inertia。
而將一個數據集中的所有簇的簇內平方和相加,就得到了整體平方和(Total Cluster Sum of Square),又叫做Total Inertia。Total Inertia越小,代表著每個簇內樣本越相似,聚類的效果就越好。因此 KMeans 追求的是,求解能夠讓Inertia最小化的質心。
KMeans有損失函數嗎?
損失函數本質是用來衡量模型的擬合效果的,只有有著求解參數需求的演算法,才會有損失函數。KMeans不求解什麼參數,它的模型本質也沒有在擬合數據,而是在對資料進行一 種探索。
另外,在決策樹中有衡量分類效果的指標準確度accuracy,準確度所對應的損失叫做泛化誤差,但不能通過最小化泛化誤差來求解某個模型中需要的資訊,我們只是希望模型的效果上表現出來的泛化誤差很小。因此決策樹,KNN等演算法,是絕對沒有損失函數的。
雖然在sklearn中只能被動選用歐式距離,但其他距離度量方式同樣可以用來衡量簇內外差異。不同距離所對應的質心選擇方法和Inertia如下表所示, 在KMeans中,只要使用了正確的質心和距離組合,無論使用什麼樣的距離,都可以達到不錯的聚類效果。
距離度量質心Inertia歐幾里得距離均值最小化每個樣本點到質心的歐式距離之和曼哈頓距離中位數最小化每個樣本點到質心的曼哈頓距離之和餘弦距離均值最小化每個樣本點到質心的餘弦距離之和
語法:
sklearn.cluster.KMeans(n_clusters=8, init='k-means++', n_init=10, max_iter=300, tol=0.0001, precompute_distances='auto', verbose=0, random_state=None, copy_x=True, n_jobs=None, algorithm='auto')
參數與介面詳解見文末附錄
例:
>>> from sklearn.cluster import KMeans>>> import numpy as np>>> X = np.array([[1, 2], [1, 4], [1, 0],... [10, 2], [10, 4], [10, 0]])>>> kmeans = KMeans(n_clusters=2, random_state=0).fit(X)>>> kmeans.labels_array([1, 1, 1, 0, 0, 0], dtype=int32)>>> kmeans.predict([[0, 0], [12, 3]])array([1, 0], dtype=int32)>>> kmeans.cluster_centers_array([[10., 2.], [ 1., 2.]])
優點
KMeans演算法是解決聚類問題的一種經典演算法, 演算法簡單、快速 。
演算法嘗試找出使平方誤差函數值最小的 個劃分。當簇是密集的、球狀或團狀的,且簇與簇之間區別明顯時,聚類效果較好 。
缺點
KMeans方法只有在簇的平均值被定義的情況下才能使用,且對有些分類屬性的資料不適合。
要求使用者必須事先給出要生成的簇的數目 。
對初值敏感,對於不同的初始值,可能會導致不同的聚類結果。
不適合於發現非凸面形狀的簇,或者大小差別很大的簇。
KMeans本質上是一種基於歐式距離度量的資料劃分方法,均值和方差大的維度將對資料的聚類結果產生決定性影響。所以在聚類前對資料(具體的說是每一個維度的特徵)做歸一化(點選檢視歸一化詳解)和單位統一至關重要。此外,異常值會對均值計算產生較大影響,導致中心偏移,因此對於"噪聲"和孤立點資料最好能提前過濾 。
KMeans演算法雖然效果不錯,但是每一次迭代都需要遍歷全量的資料,一旦資料量過大,由於計算複雜度過大迭代的次數過多,會導致收斂速度非常慢。
由KMeans演算法原來可知,KMeans在聚類之前首先需要初始化 個簇中心,因此 KMeans演算法對初值敏感,對於不同的初始值,可能會導致不同的聚類結果。因初始化是個"隨機"過程,很有可能 個簇中心都在同一個簇中,這種情況 KMeans 聚類演算法很大程度上都不會收斂到全局最小。
想要優化KMeans演算法的效率問題,可以從以下兩個思路優化演算法,一個是樣本數量太大,另一個是迭代次數過多。
mini batch 優化思想非常樸素,既然全體樣本當中資料量太大,會使得我們迭代的時間過長,那麼隨機從整體當中做一個抽樣,選取出一小部分資料來代替整體以達到縮小資料規模的目的。
mini batch 優化非常重要,不僅重要而且在機器學習領域廣為使用。在大資料的場景下,幾乎所有模型都需要做mini batch優化,而MiniBatchKMeans就是mini batch 優化的一個應用。直接上模型比較MiniBatchKMeans和KMeans兩種演算法計算速度(樣本量1,000,000)
KMeans用時接近 6 秒鐘,而MiniBatchKMeans 僅用時不到 1 秒鐘

且聚類中心基本一致
>>> KMeans.cluster_centers_array([[-2.50889102, 9.01143598], [-6.88150415, -6.88090477], [ 4.63628843, 1.97271152], [-8.83895916, 7.32493568]])>>> MiniBatchKMeans.cluster_centers_array([[-2.50141353, 8.97807161], [-6.88418974, -6.87048909], [ 4.65410395, 1.99254911], [-8.84903186, 7.33075289]])
mini batch優化方法是通過減少計算樣本量來達到縮短迭代時長,另一種方法是降低收斂需要的迭代次數,從而達到快速收斂的目的。收斂的速度除了取決於每次迭代的變化率之外,另一個重要指標就是迭代起始的位置。
2007年Arthur, David, and Sergei Vassilvitskii三人發表了論文"k-means++: The advantages of careful seeding" http://ilpubs.stanford.edu:8090/778/1/2006-13.pdf,他們開發了'k-means++'初始化方案,使得初始質心(通常)彼此遠離,以此來引匯出比隨機初始化更可靠的結果。
'k-means++'聚類演算法是在KMeans演算法基礎上,針對迭代次數,優化選擇初始質心的方法。sklearn.cluster.KMeans 中預設參數為 init='k-means++',其演算法原理為在初始化簇中心時,逐個選取 個簇中心,且離其他簇中心越遠的樣本越有可能被選為下個簇中心。
演算法步驟:
從資料即 中隨機(均勻分佈)選取一個樣本點作為第一個初始聚類中心
計算每個樣本與當前已有聚類中心之間的最短距離;再計算每個樣本點被選為下個聚類中心的概率,最後選擇最大概率值所對應的樣本點作為下一個簇中心
重複上步驟,直到選擇個聚類中心
'k-means++'演算法初始化的簇中心彼此相距都十分的遠,從而不可能再發生初始簇中心在同一個簇中的情況。當然'k-means++'本身也具有隨機性,並不一定每一次隨機得到的起始點都能有這麼好的效果,但是通過策略,我們可以保證即使出現最壞的情況也不會太壞。
'k-means++' code:
def InitialCentroid(x, K): c1_idx = int(np.random.uniform(0, len(x))) # Draw samples from a uniform distribution. centroid = x[c1_idx].reshape(1, -1) # choice the first center for cluster. k = 1 n = x.shape[0] # number of samples while k < K: d2 = [] for i in range(n): subs = centroid - x[i, :] # D(x) = (x_1, y_1) - (x, y) dimension2 = np.power(subs, 2) # D(x)^2 dimension_s = np.sum(dimension2, axis=1) # sum of each row d2.append(np.min(dimension_s)) new_c_idx = np.argmax(d2) centroid = np.vstack([centroid, x[new_c_idx]]) k += 1 return centroid
初始質心
KMeans演算法的第一步"隨機"在樣本中抽取 個樣本作為初始質心,因此並不符合"穩定且更快"的需求。因此可通過random_state參數來指定隨機數種子,以控制每次生成的初始質心都在相同位置。
一個random_state對應一個質心隨機初始化的隨機數種子。如果不指定隨機數種子,則 sklearn中的KMeans並不會只選擇一個隨機模式扔出結果,而會在每個隨機數種子下運行多次,並使用結果最好的一個隨機數種子來作為初始質心。我們可以使用參數n_init來選擇,每個隨機數種子下運行的次數。
而以上兩種方法仍然避免不了基於隨機性選取 個質心的本質。在sklearn中,我們使用參數init ='k-means++'來選擇使用'k-means++'作為質心初始化的方案。
init : 可輸入"k-means++","random"或者一個n維陣列。這是初始化質心的方法,預設"k-means++"。輸入"k- means++":一種為K均值聚類選擇初始聚類中心的聰明的辦法,以加速收斂。如果輸入了n維陣列,陣列的形狀應該是(n_clusters,n_features)並給出初始質心。
random_state : 控制每次質心隨機初始化的隨機數種子。
n_init : 整數,預設10,使用不同的質心隨機初始化的種子來運行KMeans演算法的次數。最終結果會是基於Inertia來計算的n_init次連續運行後的最佳輸出。
max_iter : 整數,預設300,單次運行的KMeans演算法的最大迭代次數。
tol : 浮點數,預設1e-4,兩次迭代間Inertia下降的量,如果兩次迭代之間Inertia下降的值小於tol所設定的值,迭代就會停下。
聚類模型的結果不是某種標籤輸出,並且聚類的結果是不確定的,其優劣由業務需求或者演算法需求來決定,並且沒有永遠的正確答案。那麼如何衡量聚類的效果呢?
簇內平方和:Total_Inertia
肘部法(手肘法)認為圖上的拐點就是 的最佳值。
# 應用肘部法則確定 kmeans方法中的kfrom scipy.spatial.distance import cdist # 計算兩個輸入集合的每對之間的距離。from sklearn.cluster import KMeansimport matplotlib.pyplot as pltimport numpy as npimport pandas as pdfrom sklearn.datasets import make_blobsplt.style.use('seaborn')plt.rcParams['font.sans-serif']=['Simhei'] #顯示中文plt.rcParams['axes.unicode_minus']=False #顯示負號X, y = make_blobs(n_samples=5000, centers=4, cluster_std = 2.5, n_features=2,random_state=42)K=range(1,10)# 直接計算ssesse_result=[]for k in K: kmeans=KMeans(n_clusters=k, random_state=666) kmeans.fit(X) sse_result.append(sum(np.min(cdist(X,kmeans.cluster_centers_,'euclidean'),axis=1))/X.shape[0])plt.figure(figsize=(16,5))plt.subplot(1,2,1)plt.plot(K,sse_result,'gp-')plt.xlabel('k',fontsize=20)plt.ylabel(u'平均畸變程度')plt.title(u'肘部法則確定最佳的K值(1)',fontdict={'fontsize':15})# 第二種,使用inertia_L = []for k in K: kmeans = KMeans(n_clusters=k, random_state=666) kmeans.fit(X) L.append((k, kmeans.inertia_))a = pd.DataFrame(L)a.columns = ['k', 'inertia']plt.subplot(1,2,2)plt.plot(a.k, a.inertia,'gp-')plt.xlabel('k',fontsize=20)plt.ylabel('inertia')plt.title(u'肘部法則確定最佳的K值(2)',fontdict={'fontsize':15})plt.xticks(a.k)plt.show();輸出結果:

但其有很大的侷限:
它的計算太容易受到特徵數目的影響。
它不是有界的,Inertia是越小越好,但並不知道合適達到模型的極限,能否繼續提高。
它會受到超參數 的影響,隨著 越大,Inertia註定會越來越小,但並不代表模型效果越來越好。
Inertia對資料的分佈有假設,它假設資料滿足凸分佈,並且它假設資料是各向同性的( isotropic),所以使用Inertia作為評估指標,會讓聚類演算法在一些細長簇,環形簇,或者不規則形狀的流形時表現不佳。
1、真實標籤已知時
可以用聚類演算法的結果和真實結果來衡量聚類的效果。但需要用到聚類分析的場景,大部分均屬於無真實標籤的情況,因此以下模型評估指標瞭解即可。
模型評估指標說明互資訊分
普通互資訊分
metrics.adjusted_mutual_info_score (y_pred, y_true)
調整的互資訊分
metrics.mutual_info_score (y_pred, y_true)
標準化互資訊分
metrics.normalized_mutual_info_score (y_pred, y_true)取值範圍在(0,1)之中越接近1,
聚類效果越好在隨機均勻聚類下產生0分。V-measure:基於條件上分析的一系列直觀度量
同質性:是否每個簇僅包含單個類的樣本
metrics.homogeneity_score(y_true, y_pred)
完整性:是否給定類的所有樣本都被分配給同一個簇中
metrics.completeness_score(y_true, y_pred)
同質性和完整性的調和平均,叫做V-measure
metrics.v_measure_score(labels_true, labels_pred)
三者可以被一次性計算出來:
metrics.homogeneity_completeness_v_measure(labels_true, labels_pred)取值範圍在(0,1)之中越接近1,
聚類效果越好由於分為同質性和完整性兩種度量,可以更仔細地研究,模型到底哪個任務做得不夠好。對樣本分佈沒有假設,在任何分佈上都可以有不錯的表現在隨機均勻聚類下不會產生0分。調整蘭德係數
metrics.adjusted_rand_score(y_true, y_pred)取值在(-1,1)之間,負值象徵著簇內的點差異巨大,甚至相互獨立,正類的 蘭德係數比較優秀,
越接近1越好對樣本分佈沒有假設,在任何分佈上都可以有不錯的表現,尤其是在具有"摺疊"形狀的資料上表現優秀,在隨機均勻聚類下產生0分。2、真實標籤未知時
輪廓係數
對沒有真實標籤的資料進行探索,常用輪廓係數評價聚類演算法模型效果。
樣本與其自身所在的簇中的其他樣本的相似度a,等於樣本與同一簇中所有其他點之間的平均距離 。
樣本與其他簇中的樣本的相似度b,等於樣本與下一個最近的簇中的所有點之間的平均距離。
根據聚類的要求"簇內差異小,簇外差異大",我們希望b永遠大於a,並且大得越多越好。單個樣本的輪廓係數計算為:
組內差異,組間差異
取值範圍越大越好。
越接近 1 表示樣本與自己所在的簇中的樣本很相似,並且與其他簇中的樣本不相似,當樣本點與簇外的樣本更相似的時候,輪廓係數就為負。
當輪廓係數為 0 時,則代表兩個簇中的樣本相似度一致,兩個簇本應該是一個簇。
語法:
from sklearn.metrics import silhouette_score# 返回所有樣本的輪廓係數的均值from sklearn.metrics import silhouette_samples# 返回的是資料集中每個樣本自己的輪廓係數silhouette_score(X,y_pred)silhouette_score(X,cluster_.labels_)silhouette_samples(X,y_pred)
例:
L = []for i in range(2, 20): k = i kmeans = KMeans(n_clusters=i) kmeans.fit(X) L.append([i, silhouette_score(X, kmeans.labels_)])a = pd.DataFrame(L)a.columns = ['k', '輪廓係數']plt.figure(figsize=(8, 5), dpi=70)plt.plot(a.k, a.輪廓係數,'gp-')plt.xticks(a.k)plt.xlabel('k')plt.ylabel('輪廓係數')plt.title('輪廓係數確定最佳的K值',fontdict={'fontsize':15})輸出結果:

輪廓係數看出,k=3時輪廓係數最大,肘部法的拐點亦是k=3,從資料集視覺化圖(文末案例)中也能看出資料集可以清洗分割3個簇(雖然初始創建了四個簇,但上面兩個簇邊界並不清晰,幾乎連到一起)。
輪廓係數有很多優點,它在有限空間中取值,使得我們對模型的聚類效果有一個"參考"。並且輪廓係數對資料的分佈沒有假設,因此在很多資料集上都表現良好。但它在每個簇的分割比較清洗時表現最好。
其它評估指標
評估指標sklearn.metrics卡林斯基-哈拉巴斯指數
Calinski-Harabaz
Indexcalinski_harabaz_score (X, y_pred)戴維斯-布爾丁指數
Davies-Bouldindavies_bouldin_score (X, y_pred)權變矩陣
Contingencycluster.contingency_matrix (X, y_pred)
這裡簡單介紹卡林斯基-哈拉巴斯指數,有 個簇的聚類而言, Calinski-Harabaz指數寫作如下公式:
其中為資料集中的樣本量,為簇的個數(即類別的個數), 是組間離散矩陣,即不同簇之間的協方差矩陣, 是簇內離散矩陣,即一個簇內資料的協方差矩陣,而表示矩陣的跡。線上性代數中,一個矩陣的主對角線(從左上方至右下方的對角線)上各個元素的總和被稱為矩陣A的跡(或跡數),一般記作。
資料之間的離散程度越高,協方差矩陣的跡就會越大。組內離散程度低,協方差的跡就會越小,也就越小,同時,組間離散程度大,協方差的的跡也會越大,就越大,因此Calinski-harabaz指數越高越好。
雖然calinski-Harabaz指數沒有界,在凸型的資料上的聚類也會表現虛高。但是比起輪廓係數,其計算非常快速。
from sklearn.cluster import KMeansfrom sklearn.metrics import silhouette_samples, silhouette_scoreimport matplotlib.pyplot as pltimport matplotlib.cm as cmimport numpy as npimport pandas as pdfrom sklearn.datasets import make_blobsX, y = make_blobs(n_samples=6000, centers=4, cluster_std = 2.5, n_features=2,center_box=(-12.0, 12.0), random_state=42)data = pd.DataFrame(X)fig, axes = plt.subplots(3, 2)fig.set_size_inches(18, 27)n_clusters = 2for i in range(3): for j in range(2): n_clusters = n_clusters clusterer = KMeans(n_clusters=n_clusters, random_state=10).fit(X) cluster_labels = clusterer.labels_ silhouette_avg = silhouette_score(X, cluster_labels) print("For n_clusters =", n_clusters, "The average silhouette_score is :", silhouette_avg) sample_silhouette_values = silhouette_samples(X, cluster_labels) colors = cm.nipy_spectral(cluster_labels.astype(float) / n_clusters) axes[i,j].scatter(X[:, 0], X[:, 1] ,marker='o' ,s=8 ,c=colors ) centers = clusterer.cluster_centers_ axes[i,j].scatter(centers[:, 0], centers[:, 1], marker='x', c="red", alpha=1, s=200) axes[i,j].set_title(f"The visualization of the clustered data when n_Clusters = {n_clusters}.") axes[i,j].set_xlabel("Feature space for the 1st feature") axes[i,j].set_ylabel("Feature space for the 2nd feature") axes[i,j].text(0,-17,f"The average silhouette_score is :nn{silhouette_avg}",fontsize=10) n_clusters += 1plt.show()輸出結果:

DBSCAN
從向量陣列或距離矩陣執行DBSCAN聚類。
一種基於密度的帶有噪聲的空間聚類 。它將簇定義為密度相連的點的最大集合,能夠把具有足夠高密度的區域劃分為簇,並可在噪聲的空間資料集中發現任意形狀的聚類。
基於密度的空間聚類與噪聲應用。尋找高密度的核心樣本,並從中擴展星團。適用於包含相似密度的簇的資料。
DBSCAN演算法將聚類視為由低密度區域分隔的高密度區域。由於這種相當通用的觀點,DBSCAN發現的叢集可以是任何形狀,而k-means假設叢集是凸形的。DBSCAN的核心元件是核心樣本的概念,即位於高密度區域的樣本。因此,一個叢集是一組彼此接近的核心樣本(通過一定的距離度量)和一組與核心樣本相近的非核心樣本(但它們本身不是核心樣本)。演算法有兩個參數,min_samples和eps,它們正式定義了我們所說的密集。較高的min_samples或較低的eps表示較高的密度需要形成一個叢集。
根據定義,任何核心樣本都是叢集的一部分。任何非核心樣本,且與核心樣本的距離至少為eps的樣本,都被演算法認為是離群值。
而主要控制參數min_samples寬容的演算法對噪聲(在嘈雜和大型資料集可能需要增加此參數),選擇適當的參數eps至關重要的資料集和距離函數,通常不能在預設值。它控制著點的局部鄰域。如果選擇的資料太小,大多數資料根本不會聚集在一起(並且標記為-1表示"噪音")。如果選擇太大,則會導致關閉的叢集合併為一個叢集,並最終將整個資料集作為單個叢集返回。
例:
>>> from sklearn.cluster import DBSCAN>>> import numpy as np>>> X = np.array([[1, 2], [2, 2], [2, 3],... [8, 7], [8, 8], [25, 80]])>>> clustering = DBSCAN(eps=3, min_samples=2).fit(X)>>> clustering.labels_array([ 0, 0, 0, 1, 1, -1])>>> clusteringDBSCAN(eps=3, min_samples=2)
eps float, default=0.5
兩個樣本之間的最大距離,其中一個樣本被認為是相鄰的。這不是叢集內點的距離的最大值,這是為您的資料集和距離函數選擇的最重要的DBSCAN參數。
min_samples int, default=5
被視為核心點的某一鄰域內的樣本數(或總權重)。這包括點本身。
更多DBSCAN聚類請參見
https://scikit-learn.org/stable/modules/generated/sklearn.cluster.DBSCAN.html#sklearn.cluster.DBSCAN
層次聚類Hierarchical Clustering 通過計算不同類別資料點間的相似度來創建一棵有層次的巢狀聚類樹。在聚類樹中,不同類別的原始資料點是樹的最低層,樹的頂層是一個聚類的根節點。創建聚類樹有自下而上合併和自上而下分裂兩種方法。
層次聚類的合併演算法通過計算兩類資料點間的相似性,對所有資料點中最為相似的兩個資料點進行組合,並反覆迭代這一過程。
簡單來說 通過計算每一個類別的資料點與所有資料點之間的歐式距離來確定它們之間的相似性,距離越小,相似度越高 。並將距離最近的兩個資料點或類別進行組合,生成聚類樹。
例:
>>> from sklearn.cluster import AgglomerativeClustering>>> import numpy as np>>> X = np.array([[1, 2], [1, 4], [1, 0],... [4, 2], [4, 4], [4, 0]])>>> clustering = AgglomerativeClustering().fit(X)>>> clusteringAgglomerativeClustering()>>> clustering.labels_array([1, 1, 1, 0, 0, 0])
層次化聚類是一種通用的聚類演算法,它通過合併或分割來構建巢狀的聚類。叢集的層次結構表示為樹(或樹狀圖)。樹的根是收集所有樣本的唯一叢集,葉子是隻有一個樣本的叢集。
聚類物件使用自底向上的方法執行分層聚類: 每個觀察從它自己的聚類開始,然後聚類依次合併在一起。連線標準決定了用於合併策略的度量。
最大或完全連線使簇對觀測之間的最大距離最小。
平均連線使簇對的所有觀測值之間的平均距離最小化。
單連線使簇對的最近觀測值之間的距離最小。
當與連通性矩陣聯合使用時,團聚向量聚合也可以擴展到大量的樣本,但是當樣本之間不加連通性約束時,計算開銷就大了:它在每一步都考慮所有可能的合併。
更多層次聚類請參見
https://scikit-learn.org/stable/modules/generated/sklearn.cluster.AgglomerativeClustering.html#sklearn.cluster.AgglomerativeClustering
附錄
n_clusters int, default=8
要聚成的簇數,以及要生成的質心數。
init {‘k-means++’, ‘random’, ndarray, callable}, default=’k-means++’
這是初始化質心的方法,輸入"k- means++": 一種為K均值聚類選擇初始聚類中心的聰明的辦法,以加速收斂。如果輸入了n維陣列,陣列的形狀應該是(n_clusters,n_features)並給出初始質心。
n_init int, default=10
使用不同的質心隨機初始化的種子來運行KMeans演算法的次數。最終結果會是基於Inertia來計算的n_init次連續運行後的最佳輸出。
max_iter int, default=300
單次運行的KMeans演算法的最大迭代次數。
tol float, default=1e-4
兩次迭代間Inertia下降的量,如果兩次迭代之間Inertia下降的值小於tol所設定的值,迭代就會停下。
precompute_distances {‘auto’, True, False}, default=’auto’
預計算距離(更快,但需要更多記憶體)。
'auto': 如果 n_samples * n_clusters > 1200萬,不要預先計算距離。這對應於使用雙精度來學習,每個作業大約100MB的記憶體開銷。
True: 始終預先計算距離。False:從不預先計算距離。
verbose int, default=0
計算中的詳細模式。
random_state int, RandomState instance, default=None
確定質心初始化的隨機數生成。使用int可以是隨機性更具有確定性。
copy_x bool, default=True
在預計算距離時,若先中心化資料,距離的預計算會更加準確。如果copy_x為True(預設值),則不會修改原始資料,確保特徵矩陣X是c-contiguous。如果為False,則對原始資料進行修改,在函數返回之前放回原始資料,但可以通過減去資料平均值,再加上資料平均值,引入較小的數值差異。注意,如果原始資料不是c -連續的,即使copy_x為False,也會複製,這可能導致KMeans 計算量顯著變慢。如果原始資料是稀疏的,但不是CSR格式的,即使copy_x是False的,也會複製一份。
n_jobs int, default=None
用於計算的作業數。計算每個n_init時並行作業數。
這個參數允許KMeans在多個作業線上並行運行。給這個參數正值n_jobs,表示使用 n_jobs 條處理器中的執行緒。值-1表示使用所用可用的處理器。值-2表示使用所有可能的處理器-1個處理器,以此類推。並行化通常以記憶體為代價增加計算(這種情況下,需要儲存多個質心副本,每個作業一個)
algorithm {「auto」, 「full」, 「elkan」}, default=」auto」
使用KMeans演算法。經典的EM風格的演算法是"full"的。通過使用三角不等式,"elkan"變異在具有定義明確的叢集的資料上更有效。然而,由於分配了額外的形狀陣列(n_samples、n_clusters),它會佔用更多的記憶體。目前,"auto" 為密集資料選擇 "elkan" 為稀疏資料選擇"full"。
cluster_centers_ ndarray of shape (n_clusters, n_features)
收斂到的質心座標。如果演算法在完全收斂之前已停止(受到'tol'和'max_iter'參數的控制),這些返回的內容將與'labels_'中反應出的聚類結果不一致。
labels_ ndarray of shape (n_samples,)
每個樣本對應的標籤。
inertia_ float
每個樣本點到它們最近的簇中心的距離的平方的和,又叫做"簇內平方和"。
n_iter_ int
實際迭代的次數。
相關文章
來源:資料STUDIO作者:Jim大量資料中具有"相似"特徵的資料點或樣本劃分為一個類別。聚類分析提供了樣本集在非監督模式下的類別劃分。聚類的基本思想是"物以類聚、人以群分",將
2021-07-22 03:03:12

「2021 AI 100 Connect Webinar」系列專場由英特爾 AI 百佳創新激勵計劃與機器之心聯合推出,專場將依託英特爾、機器之心等多方資源優勢,為企業共同探討 AI 前沿技術和產業落
2021-07-22 03:03:07

楊淨 發自 合肥量子位 報道 | 公眾號 QbitAI要不是因為在合肥,我還以為是某偉達\某特爾\某歌的釋出會。一開場,CEO抱著巨大晶圓直接就這麼上來了。這個大圓盤刻了4000顆我們自
2021-07-22 03:02:58

隨著一系列短視訊應用的大火,音視訊相關技術在業內受到越來越多的關注,與此同時,如何提升音視訊處理能力,提供更優質的媒體服務也成為亟待解決的技術問題。7 月 31 日至 8 月 1
2021-07-22 03:02:37

軟體開發公司JetBrains最近釋出了《2021年開發者生態系統現狀》報告,資料來源來自183個國家31743名開發者參與。怕你們懶,截圖個看下重點:一、受歡迎的程式語言HTML/CSS不是一
2021-07-22 03:02:25

概念遊標(Cursor)它使使用者可逐行訪問由SQLServer返回的結果集。使用遊標(cursor)的一個主要的原因就是把集合操作轉換成單個記錄處理方式。用SQL語言從資料庫中檢索資料後
2021-07-22 03:02:18